(論文)Pretrained language models
はじめに
以前にPretrained language modelsの基本について書きました。
kento1109.hatenablog.com
今回はドメイン領域(Biomedical)での固有表現認識(NER)について適用した論文をまとめます。
どんな論文?
Biomedicalから重要なエンティティ(薬品名、病名、組織名など)を抽出するタスクはかなり昔から研究されており、今でも重要なタスクです。
このドメインで抽出したいエンティティはIREXが定義している一般的なエンティティ(人名、都市名など)とは大きく異なります。下記が医療文書に出現するテキスト例で、赤いマーカーが「disease entities」、黄色いマーカーが「anatomical entities」です。

このようにBiomedicalのエンティティとなる単語はドメイン固有な場合は多く、一般的なコーパス(Wikipediaなど)ではほとんど出現しません。
そこで一般的なコーパスから訓練済モデルを構築するのではく、PubMedデータセットを用いて学習を行い精度向上を達成しました。
また、単語の分散表現をCharacter-Level CNNでも学習している点も面白いと思いました。
手法
NERモデル構造は下図の通りです。
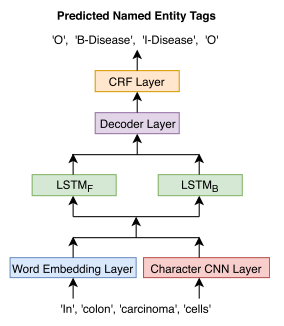
これらのレイヤーのうち、
- character-CNN
- word embedding
- BiLSTM(LSTM_F + LSTM_B)
を言語モデルで事前に学習します。
言語モデルの構造は下図の通りです。

また、NERモデルのうち、
- Decoder
- CRF
はNER固有なので、ランダムな初期値を用います。
順番に各レイヤーを見ていきます。
character-level CNN
単語を文字単位で畳み込んでその特徴量を抽出します。

単語レベルで捉えられない文字の特徴量で単語を表現できることが期待できます。
word embedding
単語レベルの特徴量を抽出します。
言語モデルでは一般的なレイヤーと言えます。
Bidirectional LSTM
character-level CNNとword embeddingの特徴量を結合したものをこの層の入力とします。


